Alpha testing fdaPDE 2.0 [cpp]¶
Cari tutti, benvenuti nella pagina di alpha-testing di fdaPDE 2.0. Scopo di questo documento è quello di mostrare quanto sia semplice tradurre uno script R o cpp, scritto per una qualunque versione precedente della libreria, usando la nuova API di fdaPDE.
Lo scopo della fase di testing è quello di rompere la libreria, e, tra le altre cose, di stressarmi. Pertanto:
se trovate bug (il codice crasha, il codice non compila, i numeri sono sbagliati, …)
se trovate qualcosa di poco intuitivo
se mancano pezzi, rispetto alla libreria attualmente su CRAN, o rispetto a lavoro pregresso che avete fatto ma che non trovate nel codice attuale
se volete aggiungere delle modelistiche che state ora sviluppando o funzionalità che trovate interessanti
segnalatemelo (mi scrivete su whatsapp o me lo dite di persona).
How to fdaPDE¶
L’idea generale è quella di scrivere degli script, che vedono fdaPDE come una libreria terza. Quindi, create un bel file main.cpp, fuori dalla cartella dove avete il sorgente di fdaPDE, e per iniziare scrivete quanto segue:
1 2 3 4 5 6 7 8 9 | |
La libreria è composta da diversi moduli. Il modo più semplice per avere un ambiente funzionante è quello di caricare l’header <fdaPDE/models.h>, il quale, attualmente, carica tutto lo stack della libreria. Per convenienza, importiamo anche il namespace fdapde.
Per compilare, al momento, solo gcc14 (o superiore) è supportato. Usate la seguente riga di codice (supponendo di essere nella cartella test/mio_test):
g++ -o main main.cpp -I../../fdaPDE-cpp -I../../fdaPDE-cpp/fdaPDE/core -O2 -march=native -std=c++20 -s
quindi esegiute con ./main.
Tipicamente, l’anatomia di uno script è la seguente:
definizione della geometria: il primo step è quello di definire la geometria del problema. In questa fase tratteremo unicamente discretizzazioni agli elementi finiti, e pertanto le nostre geometrie saranno unicamente triangolazioni. Potete caricare una triangolazione con il seguente codice:
1 2 3 | |
Alternativamente, potete generare delle semplici geometrie in maniera dinamica nel modo seguente:
1 2 3 4 | |
definizione dei dati: una volta definita la geometria, possiamo caricare i dati. A tal scopo un oggetto di tipo
GeoFramemodelizza un data frame per dati che sono spazialmente localizzati su una geometria. Potete definire unGeoFramenel modo seguente:
1 | |
L’oggetto data appena costruito è un GeoFrame definito su una triangolazione D, che rappresenta lo spazio fisico dove sono definiti i dati di interesse. data così costruito non ha alcun dato associato.
Prima di procedere, è bene sapere che GeoFrame è una struttura dati abbastanza complessa. Nella sua interezza, essa rappresenta una struttura multi-layer, ovvero una struttura in grado di gestire dati osservati potenzialmente su supporti differenti.
Tip
Potete immaginare un GeoFrame come una pila dove, alla base, abbiamo un layer fisico definito dalla geometria, sulla quale definiamo uno o più layers contenenti le osservazioni, potenzialmente osservate su supporti differenti.
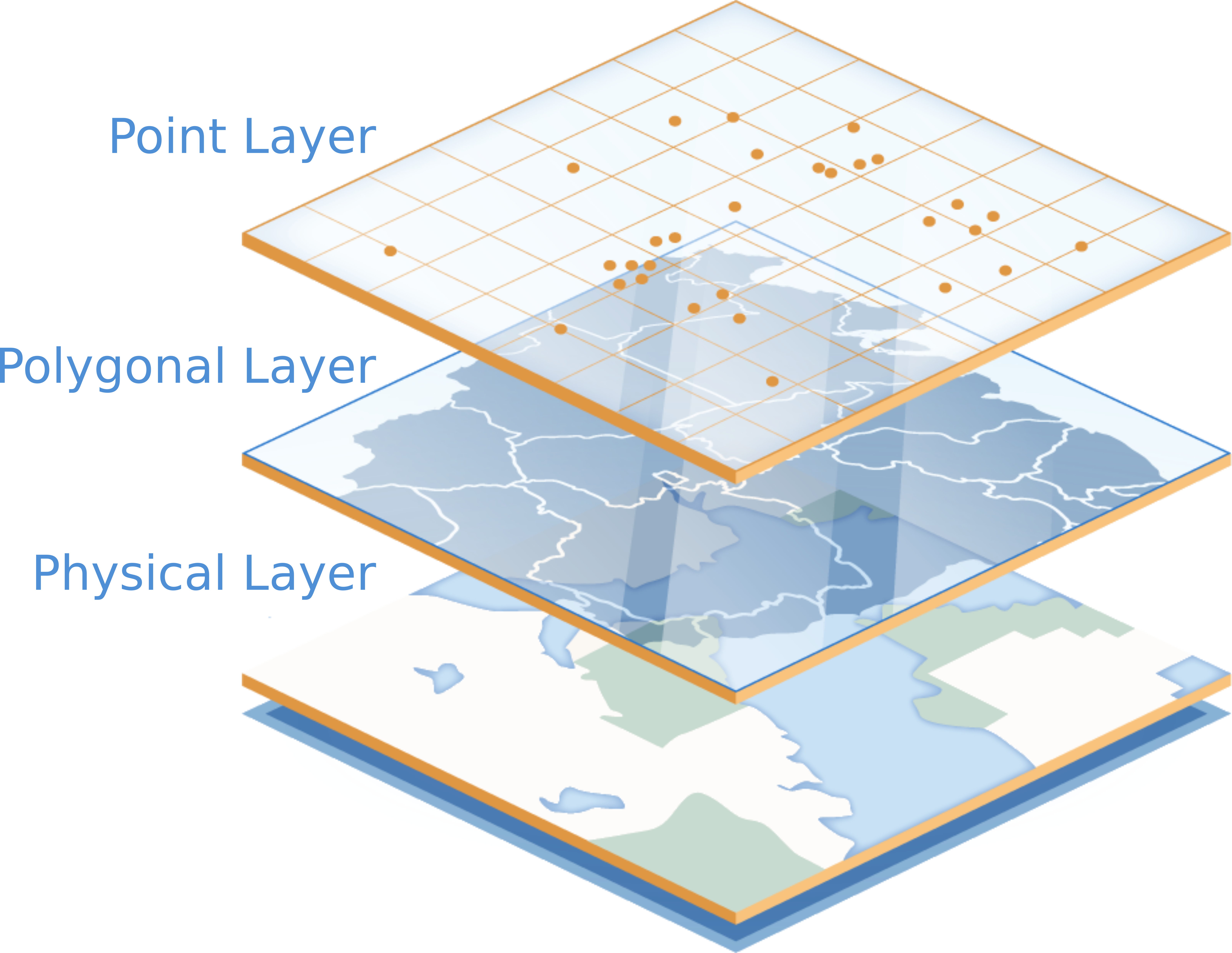
Poichè per il momento i modelli supportati gestiscono dati osservati sul medesimo supporto, i.e. sono single-layer, ed inoltre gestiscono unicamente dati scalari, ci occuperemo unicamente di questo caso.
Per aggiungere un layer scalare, ovvero in cui ad ogni locazione è associato un singolo valore numerico, si procede nel modo seguente:
1 | |
La funzione insert_sclar_layer<POINT>() inserisce un layer scalare. Per specificare che i dati sono puntuali utilizziamo il descrittore POINT. L’altro descrittore attualmente supportato è POLYGON, e definisce dati associati a poligoni, ossia quelle che per noi sono osservazioni areali.
Mentre il primo argomento di insert_sclar_layer specifica il nome simbolico del layer, il secondo argomento specifica dove i dati sono osservati. Questo può essere o il nome di un file .csv o .txt (in tal caso formattato in stile write.table) dove le coordinate sono salvate, o essere uguale al valore speciale MESH_NODES, nel qual caso i nodi della mesh sono automaticamente utilizzati come locazioni, o essere una matrice di punti definita da sorgente. Il codice seguente mostra queste ultime due casistiche:
1 2 3 4 5 | |
Per il caso di dati areali, il seguente codice definisce un layer areale con matrice di incidenza caricata da file
1 | |
La matrice di incidenza è una matrice binaria che ha tante colonne quante celle della triangolazione e tante righe quante sottoregioni. L’elemento in posizione (i, j) è 1 se la cella j-esima appartiene all’i-esima sottoregione.
Dopo aver inserito le coordinate, potete procedere all’inserimento dei dati (che devono avere la stessa numerosità del numero di locazioni). Una richiesta abbastanza frequente sarà quella di caricare dati da file, operazione che può essere realizzata con il codice seguente:
1 2 | |
I nomi delle colonne in questo caso sono presi dall’header dei file. Se avete dati generati da sorgente, è sempre possibile procedere come segue:
1 2 3 4 5 6 | |
è infine possibile visualizzare il contenuto di un layer mandando l sullo stream di output
1 2 3 4 5 6 7 8 9 10 11 12 | |
La struttura dati è in grado di eseguire operazioni molto più complesse, ma per questo tutorial ci limitiamo a questo caso base.
Per il caso di problemi spazio-temporali, GeoFrame è in grado di gestire arbitrarie tensorizzazioni di triangolazioni. Il codice seguente definisce un GeoFrame definito su un cilindro spazio-temporale:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 | |
Definite le discretizzazioni temporale T e spaziale D, GeoFrame data(D, T) definisce un geoframe sul prodotto cartesiano tra D e T. In questo caso, insert_scalar_layer<POINT, POINT>() richiede due descrittori, uno per la dimensione spaziale e uno per quella temporale. Tutte le combinazioni tra POINT e POLYGON sono supportate (permettendo, ad esempio, la gestione di osservazioni puntuali in spazio e areali in tempo (<POINT, POLYGON>) o areali in spazio e puntuali in tempo (<POLYGON, POINT>)).
insert_scalar_layer richiede quindi, oltre al nome simbolico del layer, la specifica delle coordinate fisiche effettive. In questo caso, è richiesta una coppia di valori, una per la dimensione spaziale e una per quella temporale. Nell’esempio sopra, std::pair {"locs.csv", MESH_NODES} carica le locazioni spaziali da file, mentre utilizza i nodi della triangolazione T come locazioni temporali. Tutte le combinazioni di possibilità viste in precedenza sono valide.
Tip
insert_scalar_layer<POINT, POINT>() chiamata come sopra automaticamente tensorizza le locazioni spaziali, i.e., data una griglia di punti in solo spazio (caricata in precedenza dal file locs.csv) e una griglia di punti in solo tempo, insert_scalar_layer<POINT, POINT>(...) automaticamente genera una griglia spazio-temporale di punti come prodotto tensore delle due singole griglie.
In alcuni casi questo non è un comportamento desiderabile. Questo potrebbe essere il caso se, ad esempio, le osservazioni non sono osservate su una griglia regolare, come nel setting dei processi di punto. Se si possiede una griglia di locazioni, è possibile passare direttamente la griglia nella maniera seguente
1 2 3 4 5 6 7 8 9 10 11 | |
In questo caso, poichè una griglia di punti esplicita è stata fornita tramite la matrice locs, GeoFrame non effettuerà alcuna tensorizzazione ma userà, invece, la griglia fornita. Questa opzione è possibile solo nel caso di layer <POINT, POINT>.
E possibile infine definire layers senza alcun dato associato. Questo può ritornare utile, ad esempio, nella definizione di problemi di processi di punto non marcati, dove non si ha nessuna quantità definita in corrispondenza della locazione. Questo è ottenuto semplicemente evitando di caricare alcun dato (tramite, e.g., read_csv o load_vec).
Alcuni modelli funzionali potrebbero voler lavorare su più unità statistiche simultaneamente. Questo è il caso, ad esempio, per i modelli di fPCA. In questo caso, invece di indicizzare le singole colonne del GeoFrame, è necessario individuare con un unico nome simbolico un blocco di più colonne. Questo può essere ottenuto o attraverso una chiamata a .merge<T>("nome_blocco") o caricando direttamente un blocco con .load_blk("nome_blocco", dati). Si veda il codice sottostante per un esempio:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 | |
definizione della fisica:
Tip
Non tutte i modelli richiedono una penalizzazione, pertanto questo step è opzionale.
a questo punto è possibile definire la fisica del problema. L’API cpp richiede sempre la definizione della fisica, anche nel caso semplice di penalizzazione laplaciana. Per definire la penalizzazione, definiamo le forme bilineari e lineari derivanti dalla formulazione debole del problema variazionale associato al problema di stima. Chiaramente, modelli diversi possono dare interpretazioni diverse a queste quantità, pertanto non esiste un ragionamento valido per ogni possible casistica. Indipendentemente dal modello statistico, l’API per la definizione di problemi differenziali permette la scrittura, e conseguente discretizzazione, di qualunque operatore differenziale, e di conseguenza, la risoluzione di qualunque PDE.
L’API per la definizone e discretizzazione di operatori differenziali è riportata nel seguente codice:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
FeSpace Vh(D, P1<1>); // functional space definition // trial and test function definition TrialFunction f(Vh); TestFunction v(Vh); // laplacian bilinear form auto a = integral(D)(dot(grad(f), grad(v))); // homogeneous forcing linear form ZeroField<2> u; auto F = integral(D)(u * v); // u can be any function, for instance ScalarField<2, decltype([](const Eigen::Matrix<double, Dynamic, 1>& p) { return p[0] + 2 * p[1]; // non-homoegenous forcing, here x + 2y })> u; auto F = integral(D)(u * v);Il primo passo è quello di definire lo spazio funzionale che vogliamo usare per discretizzare il problema differenziale.
FeSpacecostruisce uno spazio agli elementi finiti sulla triangolazioneD, usando elementi finiti lineari scalari (significato diP1<1>).P1<N>, conN > 1, definisce uno spazio agli elementi finiti vettoriale diNcomponenti. Gli elementi finiti di tipo Lagrange supportati arrivano fino all’ordineP5(anche se per i nostri interessi statistici non si andrà mai oltreP2).Successivamente, previa definizione delle funzioni di trial e di testing, è possibile passare alla definizione delle forme deboli. Ad esempio, la formulazione debole per un operatore di diffusione isotropa, è data come:
\[a(f, v) = \int_{\mathcal{D}} \nabla f \cdot \nabla v\]e viene tradotta in codice come
1auto a = integral(D)(dot(grad(f), grad(v)));Rimando agli esempi specifici sulle PDE per esempi più avanzati.
definizione del modello: arrivati a questo punto, abbiamo tutti gli elementi per definire la nostra modellistica statistica. Ciascun modello ha le sue specifiche, pertanto non c’è una descrizione valida per tutti i casi.
Prima di procedere dobbiamo introdurre Il concetto fondamentale di solver variazionale. Un solver variazionale è l’algoritmo per risolvere un problema del tipo:
\[\begin{split}\begin{align} & \min_{\boldsymbol{f} \in \mathbb{H}} && \mathcal{L}(\boldsymbol{f}) + \mathcal{P}(\boldsymbol{f}, \boldsymbol{f}) &&\\ & \text{s.t.} && \mathcal{C}(\boldsymbol{f}) = \boldsymbol{0} \end{align}\end{split}\]Il problema sopra indicato è fin troppo generico. Un risolutore fissa, a meno della fisica, ovvero della penalizzazione \(\mathcal{P}(\boldsymbol{f}, \boldsymbol{f})\), tutti i dettagli che definiscono il problema variazionale, e la sua risoluzione, e.g. dettagli quali la tipologia di discretizzazione usata, l’uso o meno di un approccio misto, eventuali schemi di integrazione in tempo, etc. I risolutori sono divisi per famiglia, con al momento due famiglie disponibili:
ls: least square solvers: risolvono problemi del tipo\[\min_{f \in \mathbb{H}, \boldsymbol{\beta} \in \mathbb{R}^q} \frac{1}{n} \sum_{i=1}^n (y_i - \boldsymbol{x}_i^\top \boldsymbol{\beta} - f(\boldsymbol{p}_i))^2 + \mathcal{P}(f, f)\]Tra i risolutori disponibili in questa famiglia abbiamo:
fe_ls_elliptic(a, F): risolutore ellittico con discretizzazione agli elementi finiti. Fissa\[\mathcal{P}(f, f) = \int_{\mathcal{D}} (-\text{div}[K \nabla f] + \boldsymbol{b} \cdot \nabla f + cf)^2.\]In questo caso,
adeve descrivere la forma debole dell’operatore ellittico usato nella penalizzazione, mentreFdeve rappresentare la forma lineare derivante dal termine di forzante. Il perchè di questo è da ritrovarsi nell’approccio agli elementi finiti misto usato per risolvere il problema. Si rimanda alla letteratura specifica.fe_ls_parabolic_mono(std::pair{a, F}, ic): risolutore spazio-tempo parabolico monolitico con discretizazzione agli elementi finiti. Fissa:\[\mathcal{P}(f, f) = \int_{\mathcal{D}} \Bigl(\frac{\partial f}{\partial t} -\text{div}[K \nabla f] + \boldsymbol{b} \cdot \nabla f + cf \Bigr)^2.\]adeve essere pari alla forma debole dell’operatore ellittico usato nella penalizzazione, mentreFrappresenta la forma lineare derivante dal termine di forzante.icè il vettore dell’espansione in base della condizione iniziale. Il risolutore approccia il problema in maniera monolitica.fe_ls_parabolic_ieul(std::pair{a, F}, ic, /* max_iter = */ 50, /* tol = */ 1e-4): risolutore spazio-tempo parabolico iterativo con discretizzazione agli elementi finiti in spazio e integrazione in tempo alla eulero implicitoieul. Risolve lo stesso problema dife_ls_parabolic_monoma usando un approccio diverso. A differenza dife_parabolic_mono, prende opzionalmente in ingresso i parametri di arresto dello schema iterativo.fe_ls_separable_mono(std::pair {a_D, F_D}, std::pair {a_T, F_T}): risolutore spazio-tempo separabile monolitico con discretizzazione in spazio agli elementi finiti e discretizzazione in tempo avente regolarità di sobolev maggiore di 2 (le B-Spline sono un caso specifico). Fissa\[\mathcal{P}(f, f) = \int_{\mathcal{D}}\int_T (L_{\mathcal{D}} f - u_{\mathcal{D}})^2 + \int_T \int_{\mathcal{D}} (L_{T} f - u_T)^2,\]con \(L_f\) operatore ellittico del secondo ordine in spazio e \(L_T\) operatore ellittico del secondo ordine in tempo.
{a_D, F_D}sono le forme deboli per la componente in spazio,{a_T, F_T}per quella in tempo.
de: density estimation solvers: risolvono problemi del tipo\[\begin{split}\begin{aligned} & \min_{f \in \mathbb{H}} && -\frac{1}{n} \sum_{i=1}^n f(\boldsymbol{p}_i) + \mathcal{P}(f, f) &&\\ & \text{s.t.} && \int_{\mathcal{D}} e^f = 1 \end{aligned}\end{split}\]Tra i risolutori disponibili in questa famiglia abbiamo:
fe_de_elliptic(a, F): risolutore ellittico con discretizzazione agli elementi finiti. Fissa\[\mathcal{P}(f, f) = \int_{\mathcal{D}} (-\text{div}[K \nabla f] + \boldsymbol{b} \cdot \nabla f + cf)^2.\]Il significato degli argomenti è lo stesso che si ha con
fe_ls_elliptic.fe_de_separable(std::pair {a_D, F_D}, std::pair {a_T, F_T}): risolutore spazio-tempo separabile monolitico con discretizzazione in spazio agli elementi finiti e discretizzazione in tempo avente regolarità di sobolev maggiore di 2 (le B-Spline sono un caso specifico). Fissa\[\mathcal{P}(f, f) = \int_{\mathcal{D}}\int_T (L_{\mathcal{D}} f - u_{\mathcal{D}})^2 + \int_T \int_{\mathcal{D}} (L_{T} f - u_T)^2.\]Il significato degli argomenti è lo stesso che si ha con
fe_ls_separable.
Modelli che lo richiedono possono prendere in ingresso un solver variazionale. Ad esempio, il codice seguente:
1SRPDE m("y ~ f", data, fe_elliptic(a, F));definisce un modello di regressione spaziale non-parametrico.
ynella formula (la stessa notazione di R è supportata) deve effettivamente essere un valida colonna indata. Modelli semi-parametrici possono essere gestiti manipolando la formula, ad esempioy ~ x1 + x2 + fdefinisce un modello che usa come covariate le colonnex1ex2indata.Modelli spazio-tempo possono essere definiti cambiando il tipo di solver variazionale, come indicato nel codice sotto:
regressione spazio-tempo separabile¶1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
// linear finite element in space FeSpace Vh(D, P1<1>); TrialFunction f(Vh); TestFunction v(Vh); auto a_D = integral(D)(dot(grad(f), grad(v))); ZeroField<2> u_D; auto F_D = integral(D)(u_D * v); // cubic B-splines in time BsSpace Bh(T, 3); TrialFunction g(Bh); TestFunction w(Bh); auto a_T = integral(T)(dxx(g) * dxx(w)); // bilaplacian discretization ZeroField<1> u_T; auto F_T = integral(T)(u_T * w); // modeling SRPDE m("y ~ x1 + f", data, fe_ls_separable_mono(std::pair {a_D, F_D}, std::pair {a_T, F_T}));regressione spazio-tempo parabolica, risolutore eulero implicito¶1 2 3 4 5 6 7 8 9 10 11
vector_t ic = read_csv<double>("ic.csv").as_matrix(); // physics FeSpace Vh(D, P1<1>); TrialFunction f(Vh); TestFunction v(Vh); auto a = integral(D)(dot(grad(f), grad(v))); ZeroField<2> u; auto F = integral(D)(u * v); // modeling SRPDE m("y ~ f", data, fe_ls_parabolic_ieul(std::pair{a, F}, ic));Come noto, SRPDE è solo un caso specifico di regressione spaziale. Altri modelli di regressione si comportano in maniera simile, come mostrato di seguito:
space-only non-parametric generalized regression model¶1 2 3 4 5 6
GSRPDE m( "y ~ f", data, /* family = */ poisson_distribution{}, fe_ls_ellitpic(a, F) );space-time semi-parametric separable quantile regression model¶1 2 3 4 5 6
QSRPDE m( "y ~ x1 + x2 + f", data, /* alpha = */ 0.99, fe_ls_separable_mono(std::pair {a_D, F_D}, std::pair {a_T, F_T}) );I modelli della famiglia di stima di densità, non prendono in ingresso una formula, essendo tutta l’informazione contenuta nelle locazioni spaziali. La loro definizione è altrettanto semplice:
space-only density estimation model¶1DEPDE m(data, fe_de_elliptic(a, F));space-time separable density estimation model¶1DEPDE m(data, fe_de_separable(std::pair {a_D, F_D}, std::pair {a_T, F_T}));
fit: definito il modello, il metodo
fitperforma il ftting effettivo. I parametri ricevuti dafitvariano da modello a modello.Per i metodi di regressione,
fitriceve in input i parametri di smoothing (fissati). Il numero di parametri di smoothing dipende dal solver variazionale scelto (1 per problemi solo spazio, 2 per problemi spazio-tempo).1 2
QSRPDE m("y ~ f", data, 0.99, fe_ls_elliptic(a, F)); m.fit(1e-2);Modelli di stima di densità prendono in ingresso, oltre ai parametri di smoothing, il punto iniziale dell’ottimizzazione insieme all’algoritmo di ottimizzazione utilizzato per la minimizzazione del funzionale:
1 2 3 4 5 6
DEPDE m(data, fe_de_elliptic(a, F)); m.fit( /* lambda = */ 0.1, g_init, /* optimizer = */ GradientDescent<Dynamic, BacktrackingLineSearch> {1000, 1e-5, 1e-2} );Usare
BFGS<Dynamic>come ottimizzatore avrebbe forzato la risoluzione del problema di stima di densità tramite BFGS, etc.etc.
export dei risultati: infine i risultati possono essere scritti su file per poi essere caricati, ad esempio, su R. Per esportare un file in formato csv, è sufficiente utilizzare la seguente linea di codice:
1write_csv("log_density.csv", m.log_density()); // save estimated log density in file log_density.csv
Se sei arrivato fin qui significa che sei ben motivato! Questo mostra un uso molto basic dell’API di fdaPDE. In realtà, puoi sviluppare modelli ben più sofisticati a partire dalla sola API esterna (vale a dire, senza scendere in cantina)!!
Se hai domande, sai come trovarmi :)
Di seguito trovate degli script completi di esempio:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 | |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 | |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 | |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 | |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 | |